何だか不均衡データ補正の話題は毎回tmaeharaさんからネタを頂戴している気がしますが(笑)、今回も興味深いネタを拝見したので試してみようと思います。
深層学習時代の class imbalance 対応が面白い。適当にバランシングしたデータセットで十分学習した後にフルデータセットでファインチューンするのがいいらしい。なんだこれ。
— ™ (@tmaehara) 2022年5月11日
端的にまとめると「under/upsamplingで均衡させた改変データセットで学習したNNを、改めて全データセットでfine-tuningすれば不均衡データ補正が上手くいく」という論文があるらしく、しかも割とうまくいくので採用している後発論文が少なからずあるようだ、というお話です。
tmaeharaさんが引用されていたのはこちらの論文なんですが、IEEE公式サイトのものは僕は読めないので適当にarXivで探したら以下のものが出てきました。

界隈ではtwo-phase trainingと呼ばれているようですね。基本的には画像データに対する不均衡データ多クラス分類課題で、多層CNNを適用する際に用いられる補正手法のようです。ということで、今回の記事ではこの手法がどれくらい有効かを過去記事の例を踏襲して検証してみます。
実は、この論文の検証をやるために事前に下調べをしておいたのでした。読んで字の如く、R版Kerasでfine-tuningを実装するというネタです。これで準備万端ということで、いざやってみようと思います。なお、毎回のことで尚且つ今回は特に強調しておきたいのですが、今回の検証について「ここに不具合がある」「ここが間違っている」「ここの理解がおかしい」という点がありましたら、コメントなどで是非ご指摘していただけると有難いです。
データセット
5年前に同様の不均衡データ補正手法の実験をした時に生成したのと同じデータセットを、サンプルサイズを10倍に増やしたものを今回は用意しました。というのは、NNならある程度サンプルサイズが大きい方がきちんと回ると考えたからです。これは先日のSVMとの比較実験の結果を踏まえています。
set.seed(1001) x1 <- cbind(rnorm(10000, 1, 1), rnorm(10000, 1, 1)) set.seed(1002) x2 <- cbind(rnorm(10000, -1, 1), rnorm(10000, 1, 1)) set.seed(1003) x3 <- cbind(rnorm(10000, -1, 1), rnorm(10000, -1, 1)) set.seed(4001) x41 <- cbind(rnorm(2500, 0.5, 0.5), rnorm(2500, -0.5, 0.5)) set.seed(4002) x42 <- cbind(rnorm(2500, 1, 0.5), rnorm(2500, -0.5, 0.5)) set.seed(4003) x43 <- cbind(rnorm(2500, 0.5, 0.5), rnorm(2500, -1, 0.5)) set.seed(4004) x44 <- cbind(rnorm(2500, 1, 0.5), rnorm(2500, -1, 0.5)) d <- rbind(x1, x2, x3, x42, x44, x43, x41) d <- data.frame(x = d[, 1], y = d[, 2], label = c(rep(0, 37500), rep(1, 2500))) px <- seq(-4, 4, 0.03) py <- seq(-4, 4, 0.03) x_test <- expand.grid(px, py) x_test <- as.matrix(x_test) plot(d[,-3], col = d[, 3] + 1, xlim = c(-4, 4), ylim = c(-4, 4), cex = 0.1, pch = 19)

全体としては15:1の2次元不均衡データセットです。正例領域が負例に囲まれるような形になっていて、これを外側から囲い込むような決定境界が描ければうまくいったと判定するものとします。
R版Kerasで提案手法をやってみる
以下のRコードで回してみました。同じものをGitHubに上げてあります。
library(keras) x_train <- as.matrix(d[, -3]) y_train <- as.matrix(d[, 3]) us_idx <- sample(1:37500, 2500, replace = F) x_train_us <- x_train[c(us_idx, c(37501:40000)),] y_train_us <- y_train[c(us_idx, c(37501:40000))] rnd_idx <- sample(5000) x_train_us <- x_train_us[rnd_idx,] y_train_us <- y_train_us[rnd_idx] all_idx <- sample(40000) x_train_all <- x_train[all_idx,] y_train_all <- y_train[all_idx,] model <- keras_model_sequential() model %>% layer_dense(units = 5, input_shape = 2) %>% layer_activation(activation = 'relu') %>% layer_dense(units = 7) %>% layer_activation(activation = 'relu') %>% layer_dense(units = 1, activation = 'sigmoid') model %>% compile( loss = 'binary_crossentropy', optimizer = optimizer_sgd(learning_rate = 0.08), metrics = c('accuracy') ) model %>% fit(x_train_us, y_train_us, epochs = 15, batch_size = 100) pred_class1 <- model %>% predict(x_test, batch_size = 100) plot(x_train_us, col = y_train_us + 1, xlim = c(-4, 4), ylim = c(-4, 4), cex = 0.1, pch = 19) par(new = T) contour(px, py, array(pred_class1, dim = c(length(px), length(py))), xlim = c(-4, 4), ylim = c(-4, 4), levels = 0.5, drawlabels = F, col = 'purple', lwd = 5) unfreeze_weights(model, from = 2) model %>% compile( loss = 'binary_crossentropy', optimizer = optimizer_sgd(learning_rate = 0.001), metrics = c('accuracy') ) model %>% fit(x_train_all, y_train_all, epochs = 15, batch_size = 100) pred_class2 <- model %>% predict(x_test, batch_size = 100) plot(d[,-3], col = d[, 3] + 1, xlim = c(-4, 4), ylim = c(-4, 4), cex = 0.1, pch = 19) par(new = T) contour(px, py, array(pred_class2, dim = c(length(px), length(py))), xlim = c(-4, 4), ylim = c(-4, 4), levels = 0.5, drawlabels = F, col = 'purple', lwd = 5) rm(model)

途中経過(undersamplingした時点での決定境界)がこちらなんですが、

全データでfine-tuningした結果を見てみると……まるでダメですねこれはorz ちょっと意外な結果になりました。
実はtmaeharaさんとは実験しながらやり取りさせていただいていて*1、その中で「unfreezeするlayerを出来るだけ後ろの方に寄せてみてはどうか」という話が出たので、上記のRコードの中にあるNNのlayerを増やした上で改めて後ろの方のlayerだけをfine-tuningするように変えてみたのですが、それでも全然上手くいきませんでした*2。
これがどういうことなのかは正直僕には確信がなくて、もしかしたら低次元の浅めのDNNではダメで、tmaeharaさんも指摘されていたようにある程度全体のサンプルサイズもクラス数も大きい不均衡データセットに対する「かなりlayerが多くて深い」CNNでないと機能しないのかもしれないという気もしています。いずれにせよ、このブログでやっているような2次元のpet experimentではうまくいかないようだということは言えそうです。
従来手法のundersampling + baggingでやってみる
一応、比較のために従来手法であるundersampling + baggingも同じNNでやってみます。NN部分は上記のコードから流用して、残りは以前の記事で使ったものを流用しただけです。時間節約のため、bag数は25に減らしてあります。こちらもGitHubに同じものを上げてあります。
library(keras) library(tcltk) model <- keras_model_sequential() model %>% layer_dense(units = 5, input_shape = 2) %>% layer_activation(activation = 'relu') %>% layer_dense(units = 7) %>% layer_activation(activation = 'relu') %>% layer_dense(units = 1, activation = 'sigmoid') model %>% compile( loss = 'binary_crossentropy', optimizer = optimizer_sgd(learning_rate = 0.08), metrics = c('accuracy') ) outbag.nn <- c() iter <- 25 pb <- txtProgressBar(min = 1, max = iter, style = 3) for (i in 1:iter){ set.seed(100 + i) train.tmp <- d[d$label==0, ] train0 <- train.tmp[sample(37500, 2500, replace=F),] train1 <- d[37501:40000, ] train <- rbind(train0, train1)[sample(5000),] x_train <- as.matrix(train[, -3]) y_train <- as.matrix(train[, 3]) model %>% fit(x_train, y_train, epochs = 15, batch_size = 100) tmp <- model %>% predict(x_test, batch_size = 100) outbag.nn <- cbind(outbag.nn, tmp) setTxtProgressBar(pb, i) } outbag.nn.grid <- apply(outbag.nn, 1, mean) plot(d[,-3], col = d[, 3] + 1, xlim = c(-4, 4), ylim = c(-4, 4), cex = 0.1, pch = 19) par(new=T) contour(px, py, array(outbag.nn.grid, c(length(px), length(py))), levels = 0.5, col = 'purple', lwd = 5, drawlabels = F)
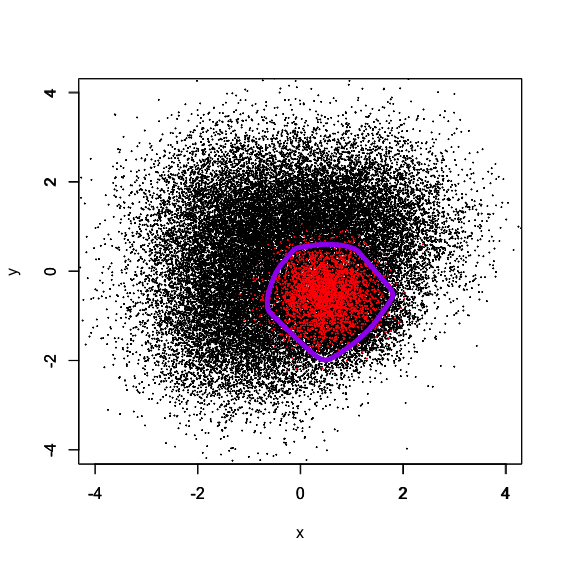
赤点で示された正例領域を綺麗に囲むように決定境界が描かれているのが分かります。
勿論これはあくまでも2次元データセットによる結果に過ぎず、より高次元なデータセットや況してや画像などの非構造化データセットでも同じことが言えるかどうかは分かりませんが、古典的なundersampling + baggingによる不均衡データ補正がNNでもきちんと回ることは確認できました。
*1:https://twitter.com/tmaehara/status/1534466202893950978
*2:ちなみに低次元なのでそもそも無闇にlayerを増やすと学習結果が不安定になるというおまけつき