この記事の前段として、まず事前に昨年書いた機械学習モデルの解釈性についての記事をご覧ください。
僕が知る限り、機械学習実践のデファクトスタンダードたるPython側ではLIMEやSHAPといった解釈手法については既に良く知られたOSS実装が出回っており、相応に実際に使ってみたというレポートも見かける状況です。一方、R側ではそこまでメインに機械学習を回す人が多くないせいか、あまりこれまで実践例を見かけないなぁと思っていました。
そんなことを考えながら先日ふと思い立ってググってみたら、意外にも幾つかの解釈手法については既にOSS実装があり、中にはCRANに上がっているものもあるのだと今更ながら知ったのでした。
ということで、二番煎じなのか何番煎じなのか分かりませんが、これらのRによる機械学習モデルの解釈法実装を今更ながら僕も試してみることにします。検証に使うデータセットは統一してUCI ML repositoryの"Wine quality"の赤ワインのデータを使います。一応、Rコード全体をGitHubに置いておきました。
いつもながらですが、今回の記事もほぼ自分向け備忘録なので特に細かい説明は大半を割愛しています。それでも誤認識や理解不足の点などあれば、コメント欄*1でお知らせくださると有難いです。
ベースラインとしての{randomForest}パッケージ
一旦、ベースラインとして古典的なランダムフォレストでvariables importance plotとpartial dependence plotを描いておきます。本来であれば"Wine quality"データセットは順序ロジットに準じた分類モデルを選択するべきなのですが、今回は面倒なので手抜きをして連続値回帰モデルでやります。
## Simple explanation by {randomForest} as a baseline d <- read.csv('https://raw.githubusercontent.com/ozt-ca/tjo.hatenablog.samples/master/r_samples/public_lib/jp/exp_uci_datasets/wine/wine_red_train.csv') library(randomForest) tuneRF(d[, -12], d[, 12], doBest = T) fit.rf <- randomForest(quality~., d, mtry = 6) varImpPlot(fit.rf) par(mfrow = c(3, 4)) for (i in 1:11){ partialPlot(fit.rf, pred.data = d, x.var = names(d)[i], xlab = names(d)[i], ylab = '', main = '') }

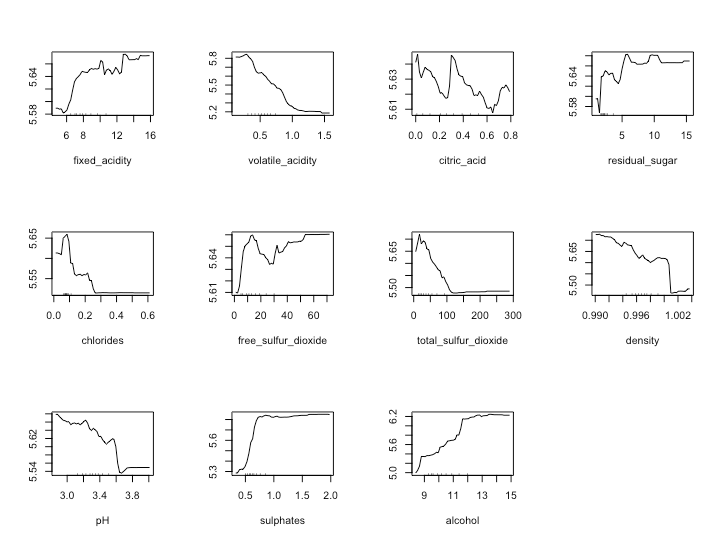
大体こんな感じになるはずです。
{interpret}パッケージ
これはPython実装の方がメインで、GitHubのrepoを見るとJuPyter notebookが色々公開されていますが、R実装についてはおまけ程度なのかマニュアルも不親切な上にvignetteもありません。よって、今回の記事では一旦スキップすることとします。ただし、見た感じではLIMEやSHAPなど一通りの解釈手法が揃っているようです。
{iml}パッケージ
こちらもCRANに上がっているパッケージです。代表的な機械学習モデルの解釈手法が(SHAPを除けば)一通り揃っています。幸いにもこちらはvignetteもあるので、単純にvignetteに沿って試してみます。ちなみにvignetteの中で扱われているのはランダムフォレストの学習モデルなので、学習部分は上のfit.rfを転用することにします。
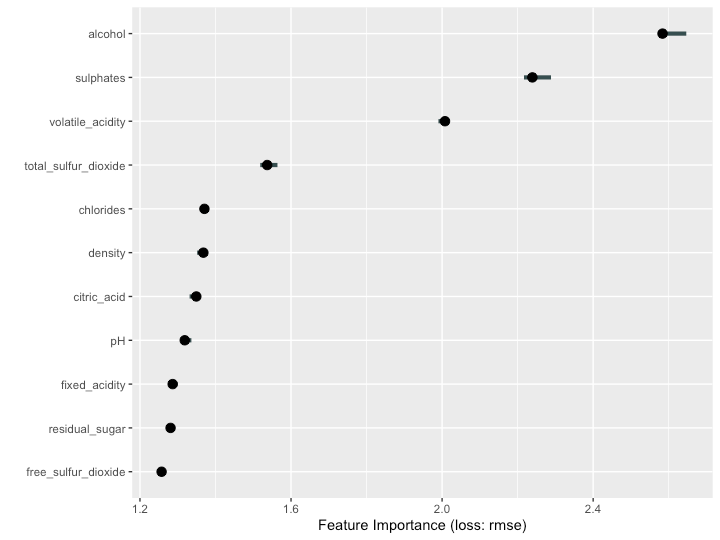
まず、feature importanceを出すところから。なお{iml}ではPredictorなどのメソッドでそれぞれのコンテナを作成して、それに対して後から色々と操作を加えていくというスタイルを取ります。
## Try {iml} # https://cran.r-project.org/web/packages/iml/vignettes/intro.html library(iml) X <- d[, -12] predictor <- Predictor$new(fit.rf, data = X, y = d$quality) # Feature importance imp <- FeatureImp$new(predictor, loss = "rmse") library(ggplot2) plot(imp)
次にfeature effects。これはpartial dependencyとほぼ同じと見て良いと思います。ここではオリジナルの{randomForest}との比較のために、ワインのテイスティングスコアqualityとの関係性がかなり非線形と思われるcitric_acid, residual_sugarを例にとってみます。
# Feature effects ale <- FeatureEffect$new(predictor, feature = "citric_acid") ale$plot() ale$set.feature("residual_sugar") ale$plot()


今度は特徴量間の交互作用について。
# Measure interactions interact <- Interaction$new(predictor) plot(interact) interact <- Interaction$new(predictor, feature = "sulphates") plot(interact) effs <- FeatureEffects$new(predictor) plot(effs)



次に、surrogate modelと言っていますが要は決定木・回帰木などシンプルなモデルへの当てはめです。
# Surrogate model tree <- TreeSurrogate$new(predictor, maxdepth = 2) plot(tree)

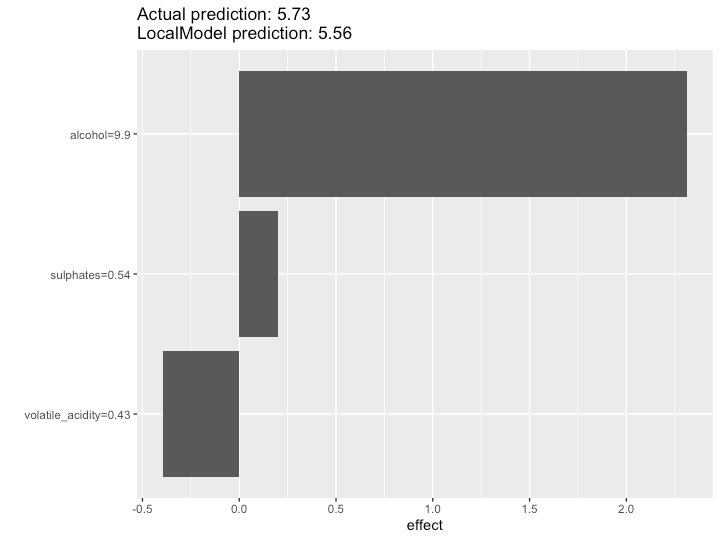
そして、冒頭のまとめ記事でも紹介したLIMEが{iml}でも扱えます。特定のサンプルの近傍に対して局所的に線形回帰モデルを当てはめ、その係数の大小関係から各特徴量の寄与度を見出そうという手法です。
# LIME: Explain single predictions with a local model lime.explain <- LocalModel$new(predictor, x.interest = X[sample(nrow(d), 1, F), ]) lime.explain$results plot(lime.explain) lime.explain$explain(X[sample(nrow(d), 1, F), ]) plot(lime.explain)

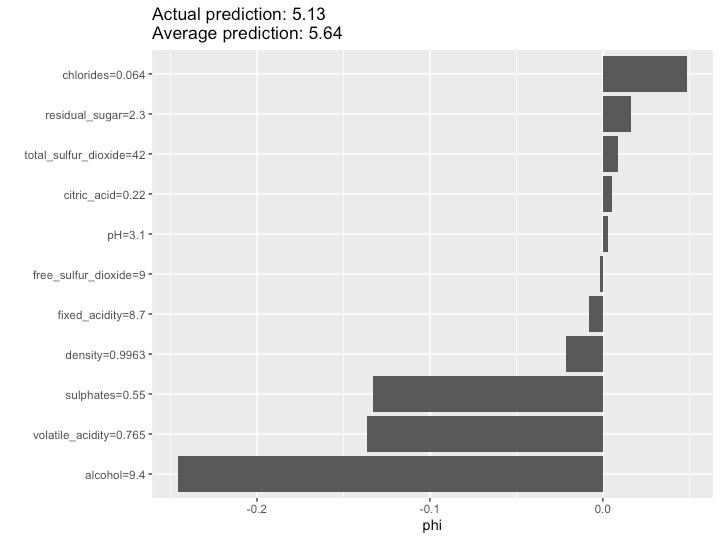
最後にShapley値の算出です。詳細は後ほどSHAP法のところで説明するとして、{iml}では以下のように実践することができます。
# Shapley: Explain single predictions with game theory shapley <- Shapley$new(predictor, x.interest = X[sample(nrow(d), 1, F), ]) shapley$plot() shapley$explain(x.interest = X[sample(nrow(d), 1, F), ]) shapley$plot()

……という感じで、一通りの変数重要度の解釈手法を試せることが分かりました。ただ、{iml}パッケージではいわゆるSHAP法に基づく変数重要度の分析ができるようではないようです*2。そこで、今度はいわゆるSHAP法的なやり方を別のフレームワークに基づいて試してみようと思います。
各種SHAP法実装
昨年の記事の中で列挙した主要な「局所的な説明」法の一つが、SHAP (SHapley Additive exPlanations) 法です。SHAPそのものについての説明は、調べた範囲では日本語だと上にリンクした記事群が分かりやすいです。極めて大雑把に言えば、SHAPとはゲーム理論における「貢献度配分」の考え方であるShapley値の考え方を、機械学習の変数重要度の算出に適用したものです。この方法だと、LIMEのように局所的に線形回帰で係数を求めるよりもさらに正確に*3、変数重要度の大小関係を求めることができるようです。
これを、サンプル一つ一つに対して当てはめるだけでなく、例えば学習データ全体に拡張すれば、ある学習モデル全体で見た場合のShapley値に基づく変数重要度(貢献度)をランダムフォレストのそれと同じ雰囲気で算出・図示することができます。これが、いわゆるSHAP法の実装に対して多くの人たちが求めていることであろうと思われます。
Pythonで有名な実装としては既にSHAPの元論文の著者自身が公開しているshapがあり、これを用いてPythonで機械学習の解釈性の問題に取り組んでいる事例は結構見かけます。一方で、Rの方ではあまり見かけないなぁと思っていたら、以下のような実装が公開されているのを見つけたのでした。
ということで、使うデータセットを"Wine quality"に変更する以外は基本的に全て公開されているチュートリアル類をなぞるだけではありますが、試してみようと思います。
pablo14/shap-valuesの実装を試してみる
これはそもそも元ネタのブログがあるので、なぞるのは簡単です。まずxgboostでモデルを学習し直します。簡単のため、パラメータ設定などは適当に決め打ちで済ませていますので悪しからず。その上でSHAP値を学習データ全体に対して算出します。
## SHAP-analysis by pablo14 ## https://github.com/pablo14/shap-values library(tidyverse) library(xgboost) library(caret) source("https://raw.githubusercontent.com/pablo14/shap-values/master/shap.R") d_mx <- as.matrix(d) # Create the xgboost model model_wine <- xgboost(data = d_mx[, -12], nround = 10, objective="reg:linear", label= d_mx[, 12]) # Note: The functions `shap.score.rank, `shap_long_hd` and `plot.shap.summary` # were originally published at https://github.com/liuyanguu/Blogdown/blob/master/hugo-xmag/content/post/2018-10-05-shap-visualization-for-xgboost.Rmd # All the credits to the author. # Calculate shap values shap_result_wine <- shap.score.rank(xgb_model = model_wine, X_train = d_mx[, -12], shap_approx = F ) # `shap_approx` comes from `approxcontrib` from xgboost documentation. # Faster but less accurate if true. Read more: help(xgboost)
あとは色々プロットするだけです。まず、SHAP値に基づいて変数重要度をプロットします。
# Plot var importance based on SHAP var_importance(shap_result_wine, top_n = ncol(d_mx) - 1)

元のshap.prep関数は上位N変数だけを抜き出して学習データ全サンプル分のSHAP値を算出できるようになっていますが、"Wine quality"データセットは11個しか説明変数がないので全部入れることにします。
# Prepare data for top N variables shap_long_wine <- shap.prep(shap = shap_result_wine, X_train = d_mx[, -12], top_n = ncol(d_mx) - 1 )
SHAP値が得られたので、あとは個々の変数ごとにSHAP値の分布を表示し、さらに変数値とSHAP値とを散布図にしたpartial dependencyっぽいものをプロットします。
# Plot shap overall metrics plot.shap.summary(data_long = shap_long_wine) xgb.plot.shap(data = d_mx[, -12], # input data model = model_wine, # xgboost model features = names(shap_result_wine$mean_shap_score[1 : (ncol(d_mx) - 1)]), n_col = 3, # layout option plot_loess = T # add red line to plot )


この2つのプロットが、SHAPといった時に多くの人が想像するものではないかと思われます。僕が見てきた範囲でも、PythonでSHAPを実践している例で出てくるのは大抵の場合はこの2つの形式のプロットです。
{SHAPforxgboost}パッケージを試してみる
実は、上のpablo14/shap-valuesの実装はこちらの{SHAPforxgboost}を下敷きにしているので、ぶっちゃけ途中までは完全に共通しています。なので、既に推定したxgboostモデルをそのまま転用した上で、{SHAPforxgboost}開発者のブログ記事に固有の部分を以下のパートではなぞっていくことにします。
まずはdependence plot。こちらの実装だと「ある変数」と「別の変数のSHAP値」との関係性を散布図にして見ることもできます。
## Try some parts of {SHAPforxgboost} ## https://liuyanguu.github.io/post/2019/07/18/visualization-of-shap-for-xgboost/ library(SHAPforxgboost) # Dependence plot g1 <- shap.plot.dependence(data_long = shap_long_wine, x = 'alcohol', y = 'alcohol', color_feature = 'sulphates') + ggtitle("(A) SHAP values of alcohol vs. alcohol") g2 <- shap.plot.dependence(data_long = shap_long_wine, x = 'alcohol', y = 'sulphates', color_feature = 'sulphates') + ggtitle("(B) SHAP values of sulphates vs. alcohol") gridExtra::grid.arrange(g1, g2, ncol = 2)
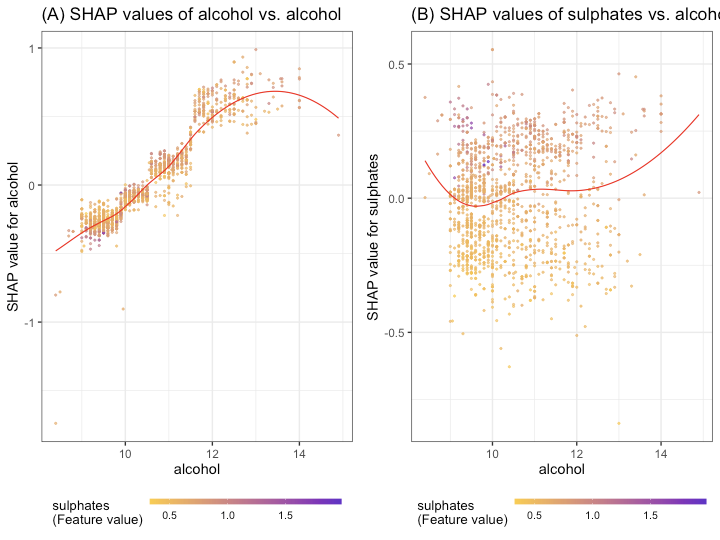
また、普通に交互作用をプロットすることもできます。
# Interaction effects shap_int_wine <- shap.prep.interaction(xgb_mod = model_wine, X_train = d_mx[, -12]) g3 <- shap.plot.dependence(data_long = shap_long_wine, data_int = shap_int_wine, x= "alcohol", y = "sulphates", color_feature = "sulphates") g4 <- shap.plot.dependence(data_long = shap_long_wine, data_int = shap_int_wine, x= "sulphates", y = "volatile_acidity", color_feature = "volatile_acidity") gridExtra::grid.arrange(g3, g4, ncol = 2)

最後はforce plotと呼ばれるもので、大雑把に言えばそれぞれの変数からの寄与度を積み上げ折れ線プロットにしたものです。最後のクラスタに分けるところはちょっと良く分かりませんでした。
# SHAP force plot shap_values <- shap.values(xgb_model = model_wine, X_train = d_mx[, -12]) plot_data <- shap.prep.stack.data(shap_contrib = shap_values$shap_score, top_n = 4, n_groups = 6) shap.plot.force_plot(plot_data, zoom_in_location = 500, y_parent_limit = c(-1.5, 1.5)) shap.plot.force_plot_bygroup(plot_data)
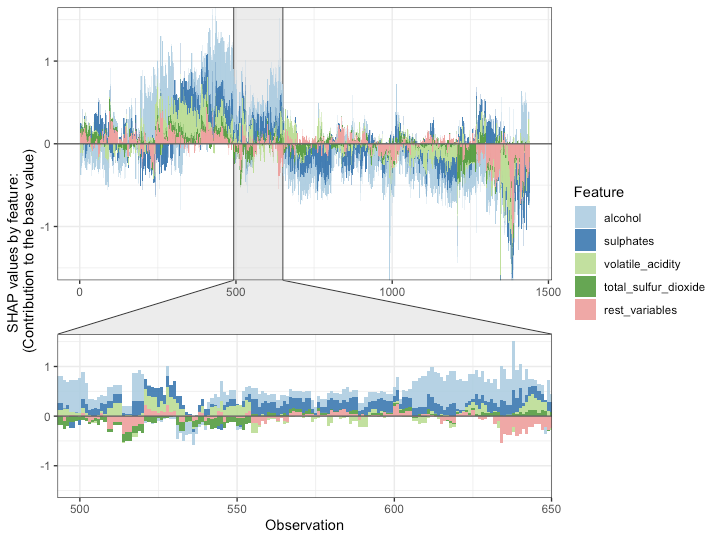

……ということで、RでもPythonライクにSHAP法を実践できることが分かりました。
感想など
正直なところ、RでPythonほどコテコテの機械学習モデルを扱うことは多くない上に、Rで「説明」(解釈)を主たる目的とする分析を行う際はほぼ例外なく「係数さえ見れば事足りる」線形モデル族か「変数重要度とpartial dependence plotを見れば十分な」ランダムフォレスト止まりなので、SHAPを初めとする最先端の解釈手法をRで実践する機会は少ないだろうと思っています。とは言え、「Rでも同じことがやれる」ということ自体に一定の価値があるのも事実なので、何かの機会があればちょっと使ってみたいです。
ちなみに、今回は簡単のためランダムフォレストとxgboostにモデルを限定しましたが、モデルの呼び出しさえうまくいけば、やり方次第では例えばKerasモデルとかでもやれるのではないかと思います。というか、樹木モデルはそもそもアルゴリズムの特性上ある程度変数重要度をまとめやすくなっているので、実際にSHAPなどの解釈手法が必要になるのはKeras以下NNベースのモデルたちでしょう。もっとも、そのためのwrapperを書くのが面倒臭そうなので、どなたか篤志家の方が書いてくれるのを待つことにします(怠慢)。